2022-04-26 12:35
作者 宋凯文

京港感染论坛
十年京港,逐梦感染病精准诊疗
前 言
2022年3月,北京大学人民医院王辉教授团队在预测抗生素耐药表型领域取得新进展。尝试绕过已知抗性机制等先验知识,预测菌株抗生素抗性表型。相关研究成果发表于《Frontiers in Microbiology》。
题目为:A Practical Approach for Predicting Antimicrobial Phenotype Resistance in Staphylococcus aureus Through Machine Learning Analysis of Genome Data(第一作者:王舒意)
▼ 背 景
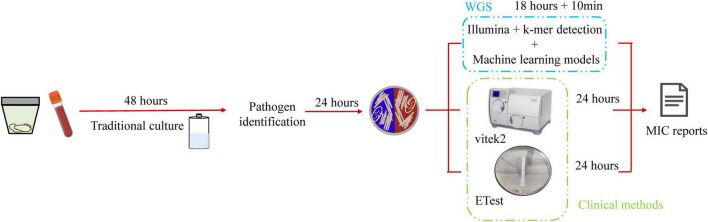
传统的微生物鉴定和药敏实验依赖于微生物培养技术,这是一个耗时的过程。经过一百多年培养技术的优化,培养时间缩短到48小时以内。然而,传统的培养技术已不能满足快速诊断日益增长的需求。尽管质谱技术现在已被广泛应用,但根据质谱判断抗生素抗性表型仍不广泛可用。近年来,基因测序技术发展迅速。最近,全基因组测序的价格已降至每株26.3美元以下,得到测序结果花费的时间可控制在18小时内。随着测序价格的降低和测序速度的加快,直接将细菌的基因型和表型联系起来就显得尤为重要。
目前,许多研究都集中在抗生素耐药性预测上,一些研究仅预测分离株是否耐药或敏感,而没有预测特定的最低抑菌浓度(MIC)值,而其他研究则更多的是基于已知耐药基因或单核苷酸多态性的存在与否预测抗生素抗性表型。
k-mer算法可以计算基因组重复区域的大小和基因组杂合度。目前,基于k-mer的研究使用的训练集数据需要相对较大,虽然较大的k值更能显示基因组特征的特异性,但这给数据采集、存储和处理带来了严重挑战,从而限制了这项技术的推广和应用。
▼ 研究过程以及结果
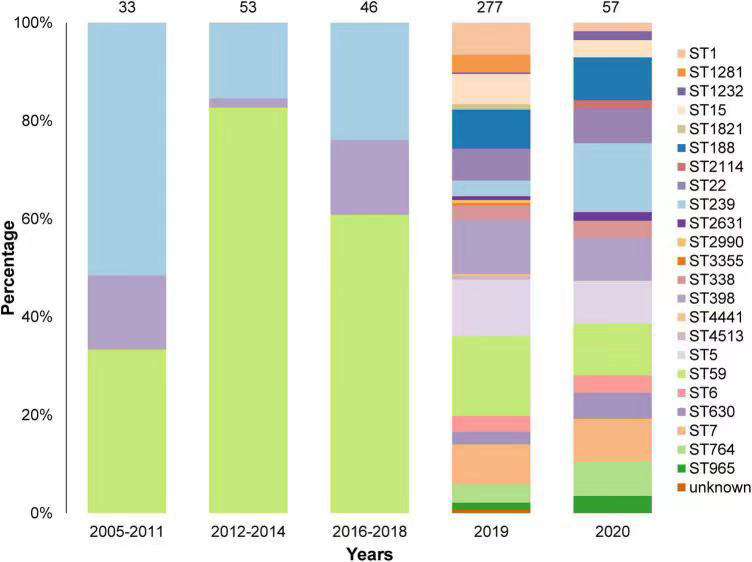
研究分析了2005年至2020年间来自中国14个省份的466株金黄色葡萄球菌(249株MRSA和217株MSSA,即对甲氧西林耐药和敏感),属于23种不同多位点序列分型,其中ST59、ST239、ST398数量最多。
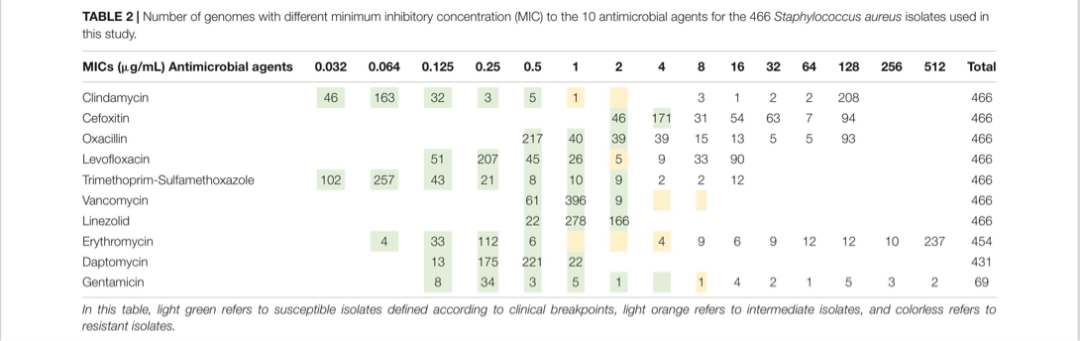
本研究对所有金黄色葡萄球菌分离株进行了抗生素敏感性测试。测试的抗生素有:克林霉素(CLI)、头孢西丁(FOX)、苯唑西林(OXA)、左氧氟沙星(LVX)、复方新诺明(SXT)、万古霉素(VAN)、利奈唑胺(LNZ)、红霉素(ERY)、达托霉素(DAP)和庆大霉素(GEN)。
分离株的MIC如下表所示,其中浅绿色是指根据临床折点定义的敏感分离株,浅橙色是指中介分离株,无色是指耐药分离株。从结果中可以看出,VAN、LNZ和DAP分离株都是全敏感的。
在收集和培养样本后,我们对所有分离株进行了全基因组测序和抗菌药物敏感性试验,以分别获得基因组和MIC数据。首先使用k-mer 算法从每个菌株的基因组中计算特征。之后使用SVM、随机森林和XGBoost三种机器学习算法来训练数据,并通过测试测试集选择的最佳模型。
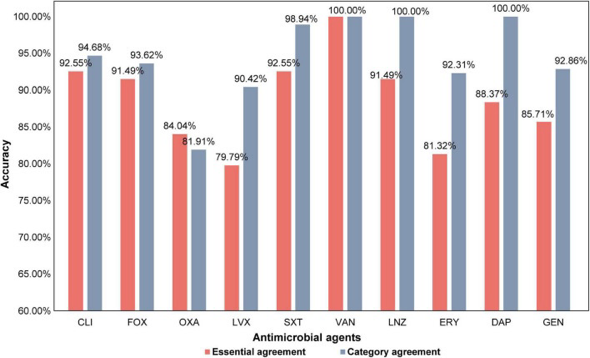
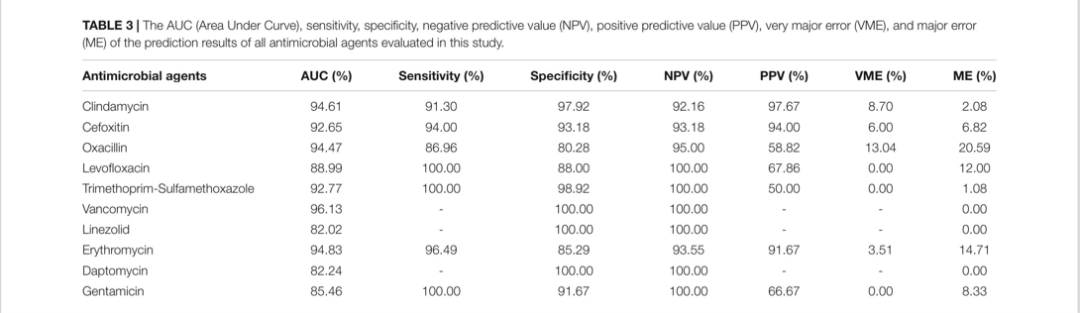
根据结果,所有抗生素的EA(基本一致性)和CA(分类一致性)几乎超过80%。除OXA、LVX和ERY外,所有抗生素的EA达到了85%以上,而CLI和SXT的EA达到了92%以上。除OXA外,所有抗生素的CA都达到90%以上,灵敏度都超过90%,特异度都超过85%。LVX、SXT和GEN的灵敏度达到100%,CLI和SXT的特异性达到97%。在这项研究中,LVX、SXT和GEN的VME低至0%。
此外,使用全基因组测序与k-mer检测和机器学习算法相结合时,抗生素耐药性预测比常规临床测试的持续时间快6小时。
▼ 小 结
本报告是一种在没有抗性基因信息的前提下,通过人工智能预测未知抗性机制菌株耐药性的方法。这是首次将k-mer与机器学习相结合预测中国金黄色葡萄球菌的最低抑菌浓度。
在临床实践过程中,医生通常根据菌株敏感性结果使用抗生素。本报告将有助于提高临床经验治疗的准确性,尤其是在无法快速获得结果的情况下。此外,在mNGS物种测定后直接分析最低抑菌浓度可以加快未来病原体诊断。
原文链接
https://www.frontiersin.org/articles/10.3389/fmicb.2022.841289/full

王舒意
北京大学人民医院医学检验学2020级博士研究生,导师为王辉教授。

宋凯文
北京大学人民医院医学检验学2022级博士研究生,导师为王辉教授。
END
作者|宋凯文
审校|余方友、陈宏斌


